近年来,深度学习逐渐成为冷冻电镜图像处理流程中颗粒挑选的常用方法。然而,现有基于深度学习的颗粒挑选方法无法在新数据训练中动态地向模型中积累新的知识。也就是说,现有模型在新样本上被训练后,虽然能够在最新的数据上获得良好的性能,但是往往无法保持其在旧数据上的颗粒挑选精度。此外,现有方法都是在特定数据集上训练出通用模型,当要加入新的训练数据时存储和计算成本都很高,大大限制了其在未曾见过的数据上的识别能力和精度。因此,我们需要改进现有深度学习网络训练的方式和方法。同时,现有的冷冻电镜设施每天都在产生大量的新数据。如果能发展一种持续学习的技术,在持续的应用过程中,让深度神经网络能够不断地学习和积累新数据中的新特征,不断地增强对生物样本图像识别能力,对发展现代化的自动化冷冻电镜系统具有非常重要的意义。
清华大学生命科学学院李雪明副教授团队,清华大学电子工程系沈渊教授团队,北京科技大学计算机与通信工程学院陈健生教授团队联合在《自然?通讯》(Nature Communications)杂志在线发表研究论文,题目为“一种用于冷冻电镜颗粒挑选知识积累的范例驱动持续学习方法EPicker”(EPicker is an exemplar-based continual learning approach for knowledge accumulation in cryoEM particle picking)。该论文报道了一种范例驱动的持续学习方法在蛋白质颗粒挑选中的应用,通过在颗粒挑选过程中不断学习新的知识来扩展检测模型识别生物大分子的能力。发展持续学习方法的重要意义在于,可以使人工深度神经网络具有类似人的学习方式,在使用中持续学习新知识、新技能,从而不断增强自身能力。EPicker经过训练之后可以挑选蛋白质颗粒、囊泡和纤维等广泛的生物对象。
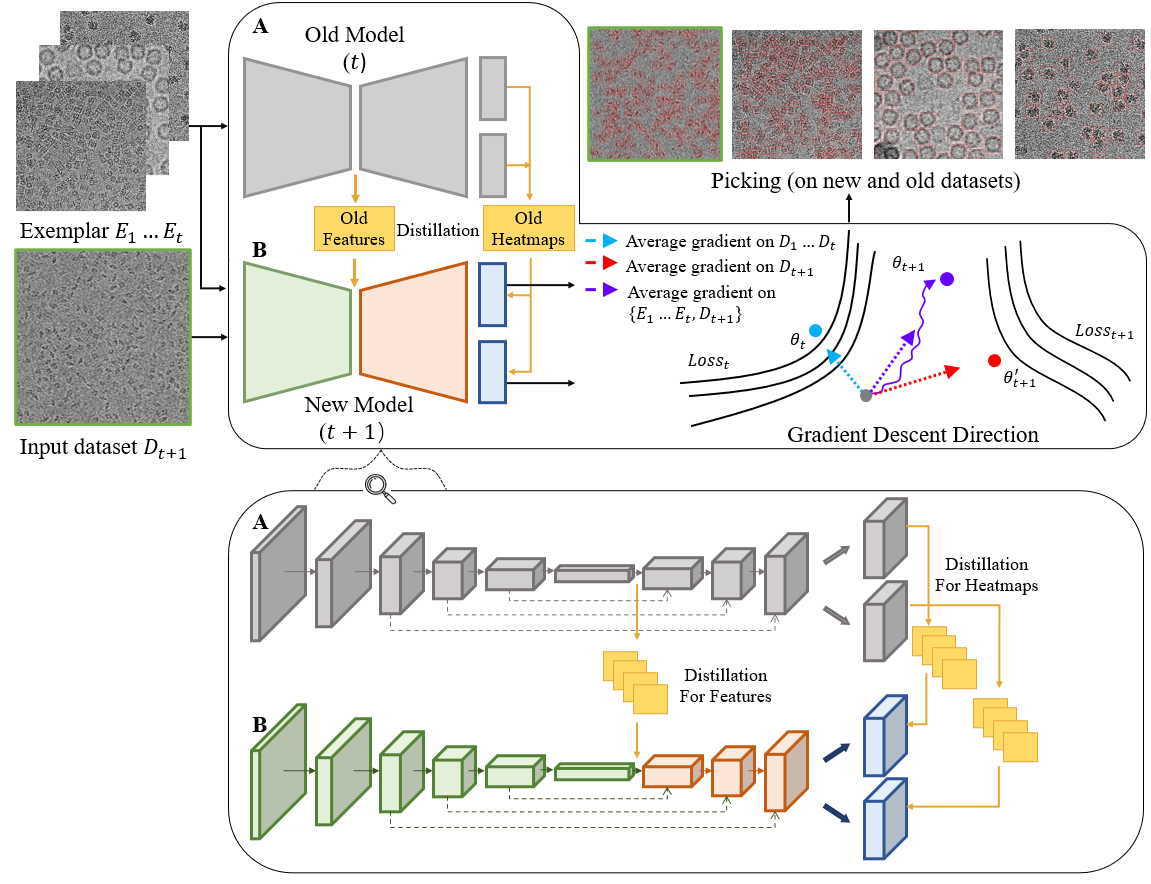
图1 EPicker的网络框架和持续学习的示意图
针对现有方法的不足之处,研究团队设计了一种基于持续学习的颗粒挑选算法,能够在训练神经网络的过程中不断积累新的颗粒挑选知识,提高通用模型的颗粒挑选能力。该算法通过设计双路网络结构(图1)和融合知识蒸馏、历史回放、正则化、稀疏标注方法,将新样本的知识不断积累到通用模型中去的同时,不会遗忘旧知识。这很好解决了模型在新数据上训练后无法挑选旧数据样本的问题。基于这些算法,研究团队开发了一个名为EPicker的新软件系统。为了进一步扩展方法的适用范围,团队针对广泛的生物对象设计了相应的挑选算法,包括挑选囊泡和纤维等多种不同的生物对象,支持有偏和无偏的颗粒挑选方式以满足用户的不同需求等等。通过在具有代表性和挑战性的数据集上进行大量实验,并与目前较为流行的颗粒挑选方法进行对比,验证了EPicker的有效性和优越性(图2)。实验结果表明,EPicker可以通过高效、高度自动化的持续学习过程得到精度高、召回高且泛化能力强的蛋白质颗粒挑选结果。
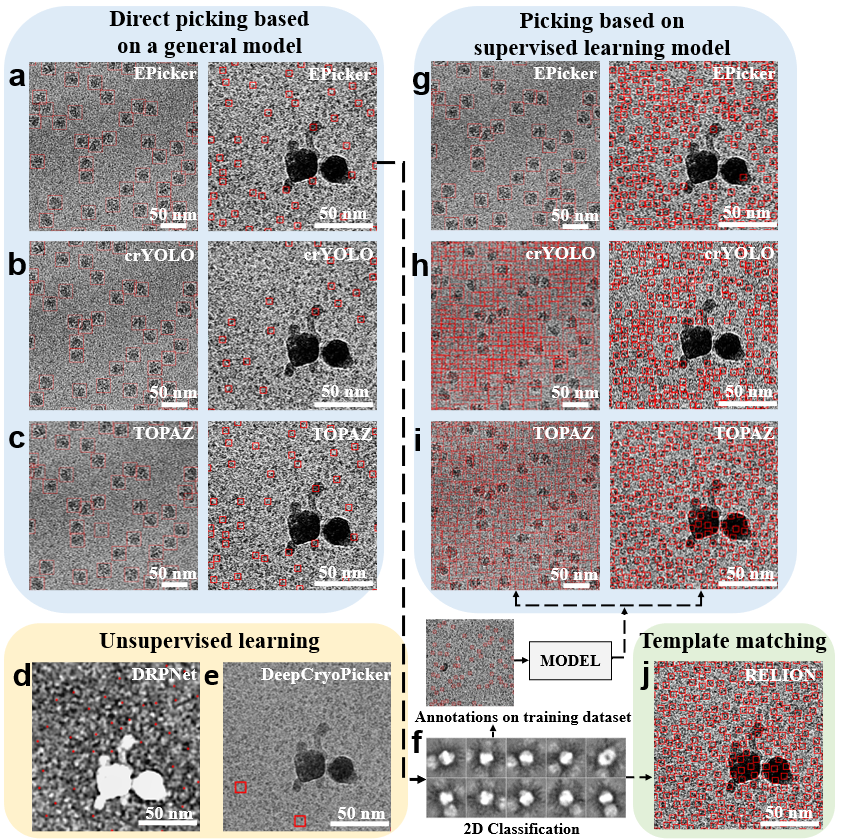
图2 不同方法的颗粒挑选结果比较
清华大学生命科学学院李雪明副教授、清华大学电子工程系沈渊教授,北京科技大学计算机与通信工程学院陈健生教授为本文的共同通讯作者。清华大学电子工程系2019级硕士生张馨予,2020级硕士生赵天放为该论文的共同第一作者。本工作获得了科技部重点研发计划,国家自然科学基金委,北京市结构生物学高精尖创新中心,北京市生物结构前沿研究中心,生命科学联合中心和北京信息科学与技术国家研究中心等的资金支持。